TaqMan SNP基因分型
TaqMan SNP基因分型
TaqMan®SNP基因分型技术利用Taq聚合酶的5'核酸酶活性在PCR期间产生荧光信号。对于每个SNP,该分析使用仅在SNP位点序列不同的两种探针,一种探针与野生型等位基因互补,另一种与变异等位基因互补。该技术利用FRET技术,将5'报告基因染料和3'淬灭染料共价连接至野生型和变异等位基因探针。当探针完好无损时,荧光会被抑制,因为淬灭染料位于报告染料的附近。在PCR退火步骤中,探针与目标SNP位点杂交。在PCR延伸过程中,由于Taq聚合酶的5'核酸酶活性,导致报告染料和猝灭染料被释放,从而导致报告染料的特征荧光增强。核酸外切酶活性仅在完全杂交的探针上发生,因为含有错配碱基的探针不会被Taq聚合酶识别。
在PCR反应结束时,将测量两种报告染料的荧光信号。信号的比率将指示样品的基因型(见图)。
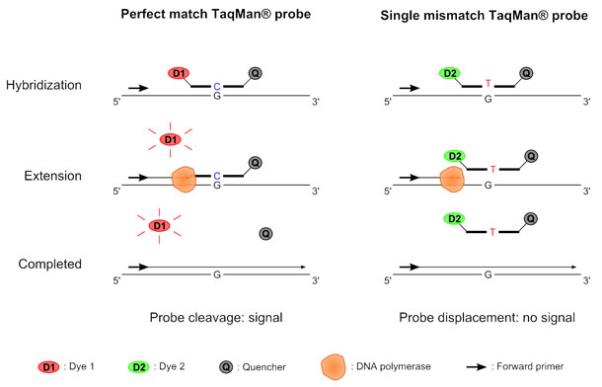
在大多数测定中,使用第三种染料(例如ROX)的信号对两种报告染料的荧光信号进行标准化,其强度与模板DNA浓度和PCR反应程度成正比。通常,报告染料信号在图中可见。许多相关的基因分型系统都采用了报告猝灭剂技术(例如Invader®,MolecularBeacons®,Scorpion®和其他探针技术),并且可以使用SNP基因分型插件以与TaqMan®探针相同的方式进行分析和可视化。
BioNumerics中的SNP分析
BioNumerics SNP识别插件为可靠的SNP识别和基因分型提供了一个全自动平台。该插件非常灵活,可以适应TaqMan SNP基因分型数据分析的不同工作流程:
1. 在其他软件中执行自动识别,然后将识别及其相应的置信度值导入BioNumerics中。
2. 在Bionumerics导入过程中执行自动识别。
3. 将数据导入为“未识别”,然后在SNP识别窗口中执行自动识别。
通过其丰富的集成环境,BioNumerics提供了许多其他软件工具无法提供的功能:
l 强大的数据库基础,包括用户和安全功能以及符合最高标准的可选审计追踪功能。
l 一个用于分析SNP基因型数据的平台,以及许多其他技术,例如HDA / CSCE、MLPA、微卫星、序列分析等。
l 从导入原始数据到报告结果和问题样本的自动化程度无可比拟。此外,该软件可使用Python进行完全脚本化,以实现各种定制化操作。因此,BioNumerics非常适合高通量分析。
l BioNumerics提供了多种数据挖掘、分析和统计工具,可对大型数据集进行聚类、统计分析和假设检验。
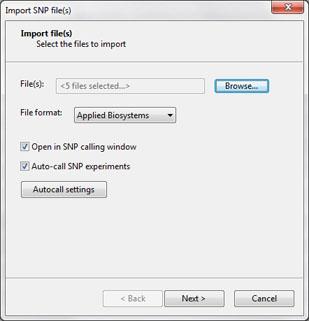
导入SNP数据
目前,该插件支持五种不同的文件格式(可以根据要求添加其他格式):
l Applied Biosystems 7900HT Fast Real-Time PCR System: 包含已处理的384孔板读数的TXT文件
l BMG Labtech 384孔微孔板读取器:包含微孔板中的原始荧光值的DAT文件
l 由Douglas Scientific Array Tape system生成的格式为384孔的TXT文件。
l Fluidigm Dynamic Array, 包含多达9,216个数据点(针对96个SNP测试了96个样本)的CSV文件
l 由Tecan Safire或Tecan Infinite微孔板读取器生成的TXT文件,为96孔和384孔微孔板格式
可以批量导入多个文件,并且可以选择在导入过程中软件执行自动SNP识别。
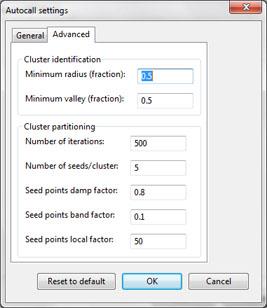
自动识别SNP
自动识别SNP算法是插件中最关键的元素,并决定了在高通量环境中有效且成功的SNP基因分型。BioNumerics使用基于种子的迭代分区算法来根据现有基因型识别SNP聚类。自动识别设置分为两个选项卡,分别代表SNP分配的常规设置和阈值(左图)以及分区算法的高级设置(右图)。 尽管默认分区设置通常表现出色,但可以针对任何特定数据源进行优化。该算法还可以在多倍体基因组中成功识别SNP(从理论上说是七倍体)。
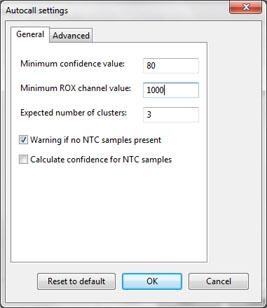
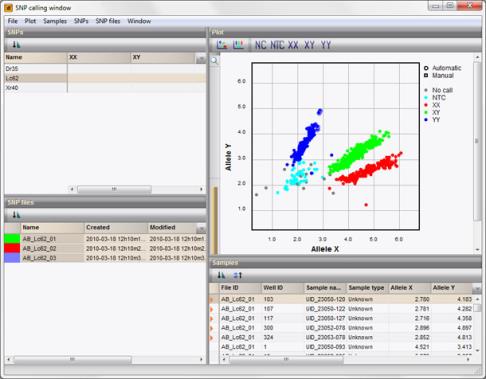
SNP识别窗口
可以在SNP识别窗口中打开单个或多个SNP文件。左上面板列出了在所选文件中找到的所有SNP,可以从中选择单个SNP以显示在绘图窗口中。SNP识别还可以按文件或针对选定文件(左下面板)绘制。
在图中,可以使用套索选择工具选择识别的SNP。
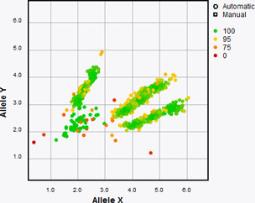
默认情况下,SNP识别根据定义的接合性识别、NTC识别和“未识别”(上图)以不同的颜色显示。这些颜色可以由用户更改。识别的统计置信度也可以用作绘图颜色的基础(右)。在第三种视图中,颜色是基于SNP文件显示。
SNP识别可以通过其置信度,ROX信号或其他参数进行排序和过滤。
可以使用直角坐标、极坐标和对比度坐标(如下所示)显示该图。
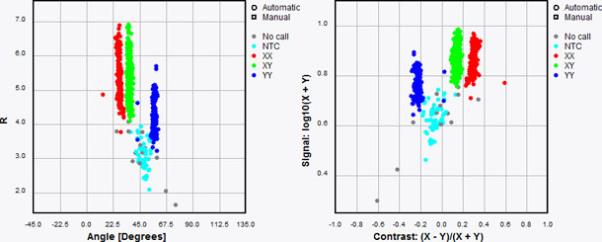
用户可以更改所选的SNP识别。 对于每个识别的SNP,软件都会显示是手动分配还是自动分配。可以导出带有识别和置信度信息的制表符分隔的文件,以便在数据库中选择任何样本。
分析和基因分型
BioNumerics中的分析不以SNP的识别结束。该软件为数据挖、聚类、筛选、识别和统计分析提供了许多高级工具。丰富的分析平台与强大的数据库功能相结合,使BioNumerics非常适合在实验室范围内或实验室间进行长期基因分型的项目。最重要的是,BioNumerics还为各种其他基因分型技术提供了同样强大的工具,包括AFLP谱带评分、MLPA分析基因组测序和SNP分析等。不同技术的结果可以各种方式组合起来,以获得更确定的分析。
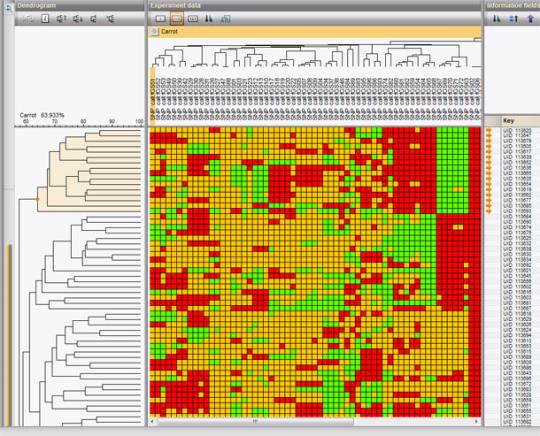
下图是胡萝卜样本和SNP的双向聚类示例,很好地说明了一方面样本组与另一方面SNP组之间的相关性。
可以使用高级查询生成器在整个项目和数据库中筛选具有特定基因型(合子性的组合)的样本。 为了实现更高水平的筛选和自动化功能,可以构建决策网络。
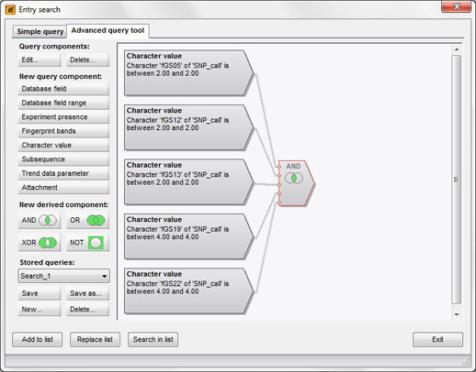
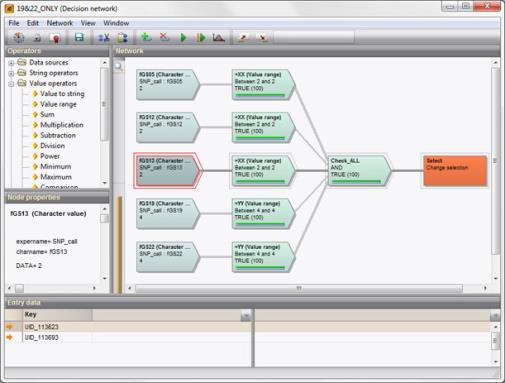
该应用程序的实现是通过BioNumerics中基于许可证的SNP调用插件实现。请与供应商联系以获取许可信息。


中国区官方授权总经销商
以上资料由BioNumerics中国区官方授权总经销商独家提供,如需转载请主动标明出处。
客服专线:010-53639817 53639871
市场专线:15712982811 13552183571
QQ客服:3545869936 3316774663
搜索关键词:
比利时Applied-Maths Bionumerics、比利时Bionumerics、BioNumerics生物信息分析软件、BioNumerics生物分析软件、BN分析软件、PFGE分析软件、Bionumerics软件7.6、Bionumerics软件6.6、食源性疾病、